干货 | ClinGen对使用ACMG指南中PVS1证据的建议
时间:2020-01-13 10:48:24 来源:华大基因学院 浏览数:29738
摘 要
2015年美国分子遗传协会(ACMG)协同美国分子病理学会(AMP)共同发布了临床数据解读与报告的标准指南,为序列变异解释提供了框架。
该指南对变异位点进行了5个等级的分类,分别为致病、疑似致病、临床意义未明、疑似良性、良性,并提供了28个标准,每个标准对应了一个代码(如 PVS1 , BA1)代表不同类型的证据,每个标准的代码由两部分组成,致病性和强度,致病性又分为良性(B)及致病(P),证据强度由强到弱依次分为单独证据(A),非常强(VS),强(S),中等(M), 支持(P) ,除此之外指南提供了相应的组合规则对突变位点进行判定。
指南中仅有的一个极强的致病证据(PVS1),具体解释为“当一个疾病的致病机制为功能丧失(LOF ,包括无义突变,移码突变,经典�1或2的剪接突变,起始密码子变异,单个或多个外显子缺失),若变异位点为功能丧失突变,则适用PVS1”, PVS1与一个中等致病证据(PM)组合即可判定为疑似致病突变,可见此种证据的重量,然而该指南没有对不同的功能缺失类型进行详细说明,实际执行过程存在种种的困难,造成不同的人员对相同的位点给出不一致的解释。
ClinGen序列变体解释(SVI)工作组提出了新的补充,有助于对预测功能变异丧失的致病标准(PVS1)进行一致和准确的解释。
内容可总结为以下两个方面
No.1 证据等级的重新划分
ACMG指南对PVS1证据定义的同时提到并非所有LoF变异强度都相同,并列举了一些需要注意的特殊情况,这些情况下不能认定为PVS1,但并未对这些情况的证据等级情况进一步说明;SVI根据LOF变异影响程度的不同将极强的致病证据(PVS1)重新划分为4个等级,分别为PVS1(PVS)、PVS1_Strong
(PS)、PVS1_Moderate(PM)和 PVS1_Supporting(PP),PVS1证据等级水平可以根据每种变异类型的强度而变化。
No.2细化的判定标准及流程
"疾病的致病机制为LoF"的判定标准
ACMG指南提到“PVS1仅适用于LoF是疾病致病机制的情况下”但并未提供判定的标准,SVI对此提供了一个框架性的标准。
标准规定了4个不同的等级,每个等级对应不同的“加权系数”用于加权PVS1判定流程的结果(图1)。
另对于常染色体显性遗传方式遗传的疾病的基因即使对于具有明确或强有力的疾病关联证据的基因,也不一定建立疾病机制(单倍剂量不足,功能获得或显性负性效应)的情况,SVI提供了一些资源帮助评估此种特殊情况,如 ClinGen haploinsufficiency (HI) 评分及EXAC 及GnomAD提供的PI值和o/e值。
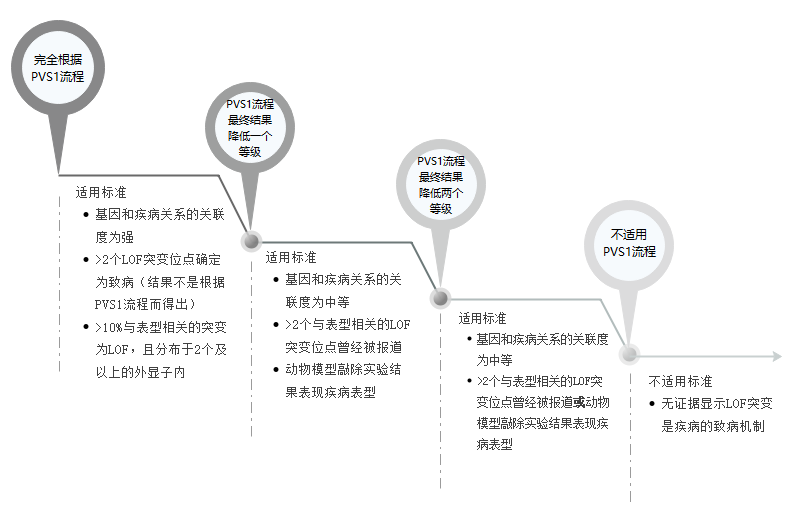
图1:基因和疾病关联度等级判定标准
PVS1判定流程的细化
SVI从不同的维度(nonsense, frameshift,canonical �1 or 2 splice sites,duplication等等)出发结合在执行上具体的考虑因素(多条转录物和无义介导的mRNA降解(NMD)、开放阅读框是否改变等)对31中不同的情况进行了致病性的判定。(图2-图4)
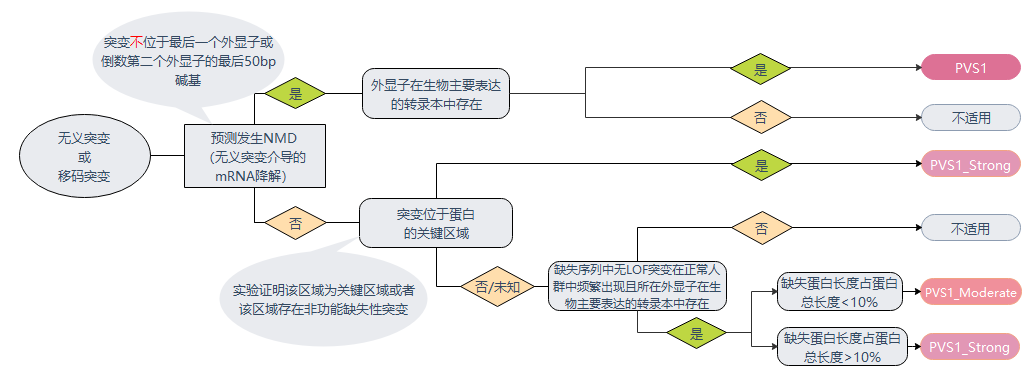
图2:无义突变或移码突变判定流程图
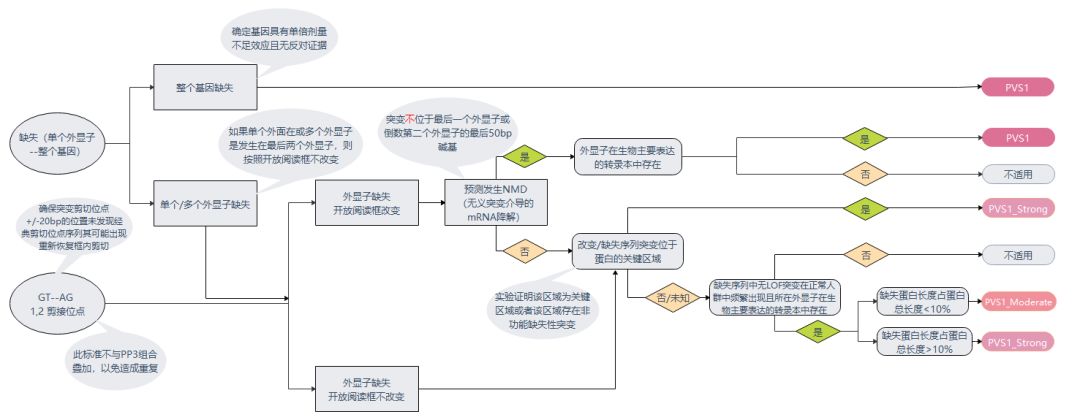
图3:缺失或经典剪切位点突变判定流程图
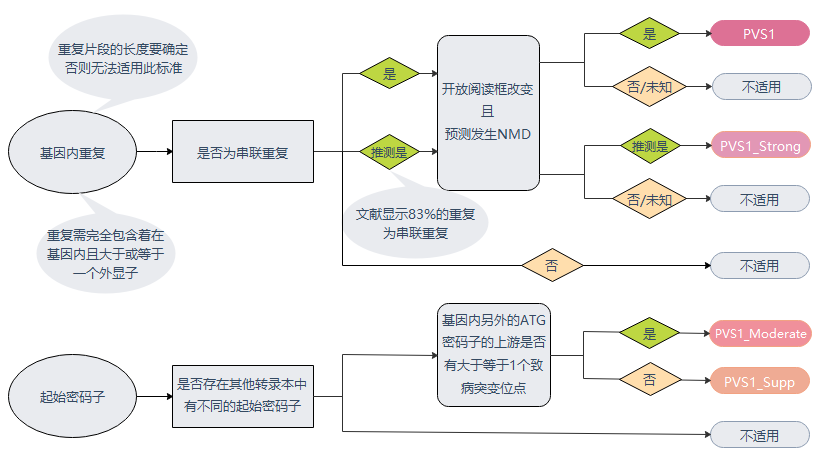
图4:基因内重复或起始密码子突变判定流程图
总的来说, SVI把2015版ACMG / AMP指南在PVS1中提到的内容进行了更细致的划分及定性,并补充了部分缺失的内容,具有更强的操作性,一定程度上降低了主观因素的影响,结果更加精准。
- 上一篇:【干货分享】R 包初学者指南






 工信部网站备案号:
工信部网站备案号: